定量ターゲットプロテオーム解析技術
松田 史生
大阪大学大学院情報科学研究科


個別技術紹介
松田 史生
大阪大学大学院情報科学研究科


スマートセルが発現している数十種のタンパク質の発現量を高感度に一斉定量する手法を開発した。大腸菌、出芽酵母、コリネ菌、油脂酵母などの中心代謝酵素タンパク質のMRMアッセイメソッド作成が完了した。スマートセルプロジェクトで作成したMRMアッセイメソッドをデータベース化した。
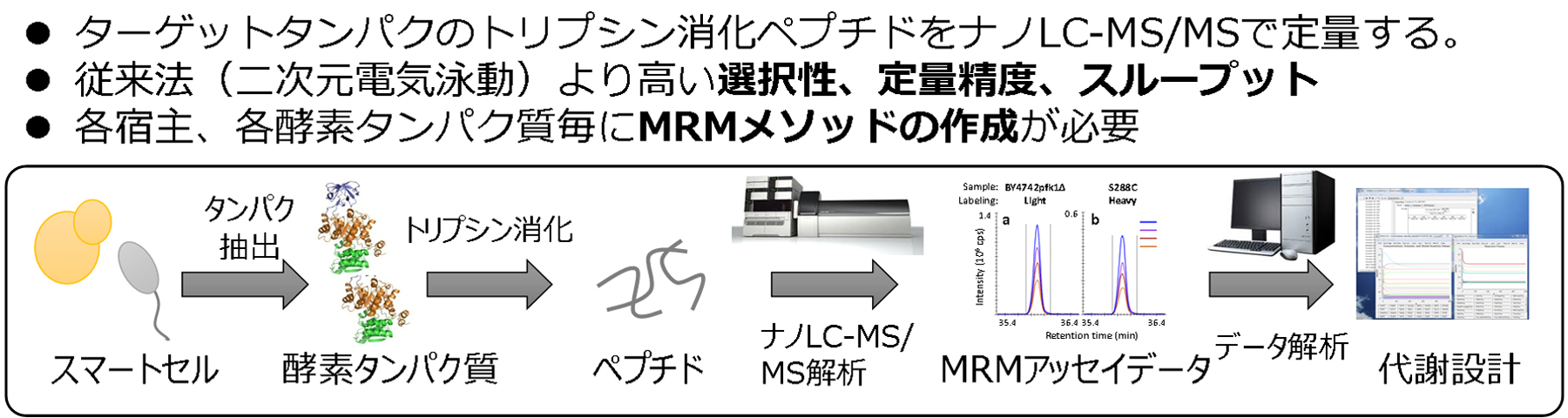
スマートセルプロジェクトでは、細胞が持つ物質生産能力を最大限に引き出すために、代謝経路の合理的な改変が求められる。代謝経路の改変では宿主微生物のゲノムを人為的に書き換え、酵素タンパク質存在量を増減させる。細胞内のタンパク質量には、転写、翻訳、タンパク質分解に関わる様々な要因が関わるため、デザイン通り酵素タンパク質量が増減できたか迅速に評価する計測手法が必要となる。そこで、トリプシン消化ペプチド混合物を液体クロマトグラフで分離し、トリプル四重極型質量分析装置の選択反応モニタリングモード (MRM) モードで分析する高精度定量ターゲットプロテオミクス法が、スマートセルの評価に最適であることに注目した。島津製作所と共同で純国産ターゲットプロテオミクス分析システムの開発を進めている(図1)
サンプル前処理の出発点は、50μgのトータルタンパク質を含む100 μl程度の粗タンパク質抽出液である。これを還元アルキル化後、トリプシン消化を一晩行い、得られたトリプシン消化ペプチドは固相抽出法を用いて脱塩する。出芽酵母用の前処理法が、油脂酵母、大腸菌、コリネ菌といったさまざまな有用微生物において有効であることを確かめた。データ取得にはナノLC-MS/MS(島津製作所LCMS-8060)を利用している。一般的な20-30サンプル程度の分析プロジェクトを2日程度で終了できるスループットを実現している。
大腸菌、出芽酵母、コリネ菌、油脂酵母などの中心代謝酵素タンパク質のMRMアッセイメソッド作成が完了している。また作成したMRMアッセイメソッドをデータベース化し、必要に応じてカスタマイズできる基盤構築を進めている。
数十種のタンパク質の発現量を高感度に一斉定量することができます。
抗体の作成が必要ありません。
1) F. Matsuda et al. : Mass Spectrometry, 6(1), A0056 (2017)
最終更新日:2022年11月14日 11:44